티스토리 뷰
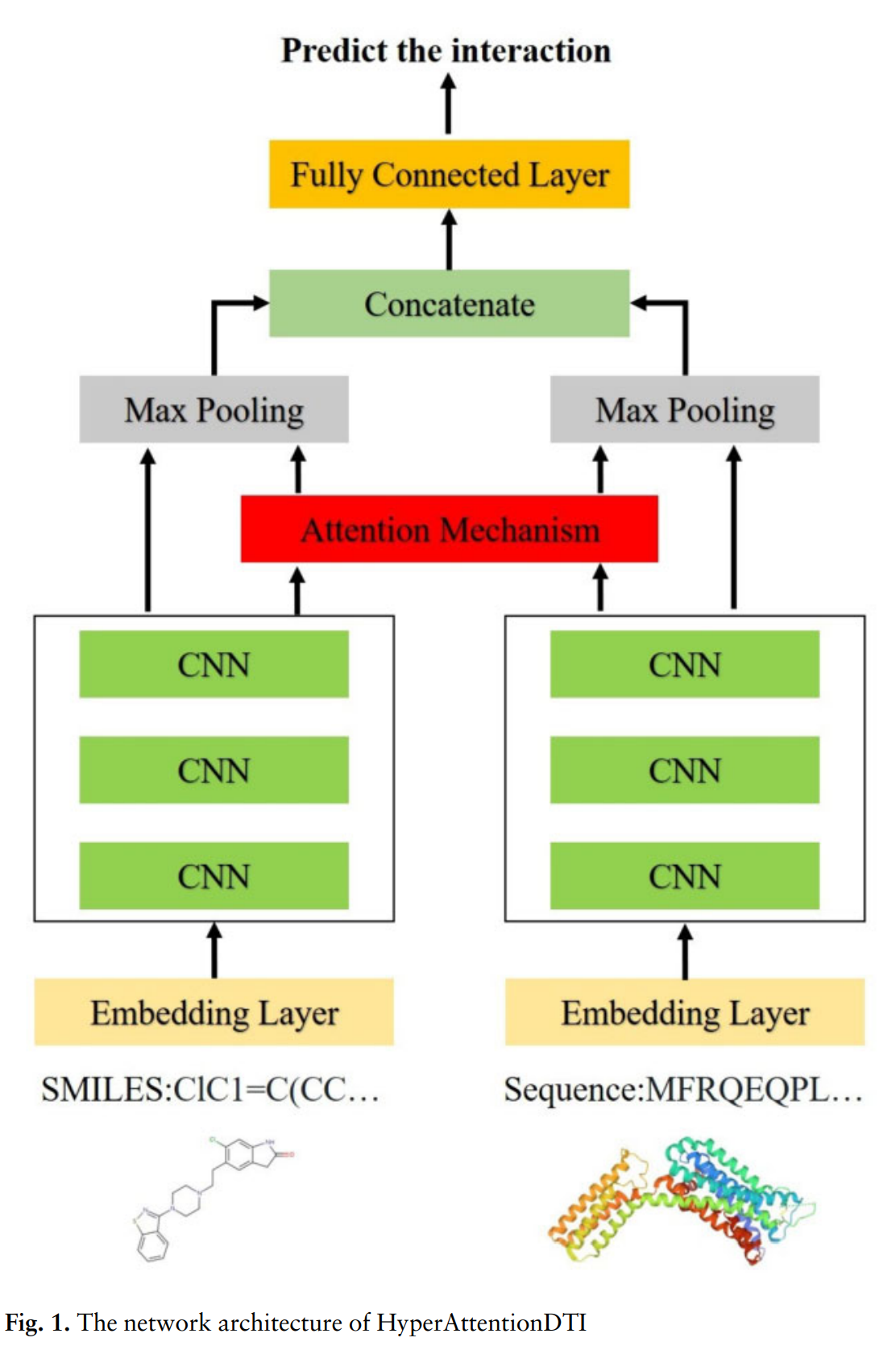
간단 요약
1. 모델이름 : HyperAttentionDTI
2. 저널 : Bioinformatics(IF 6.0 ~ 7.0)
3. Published date : 2022 / 02
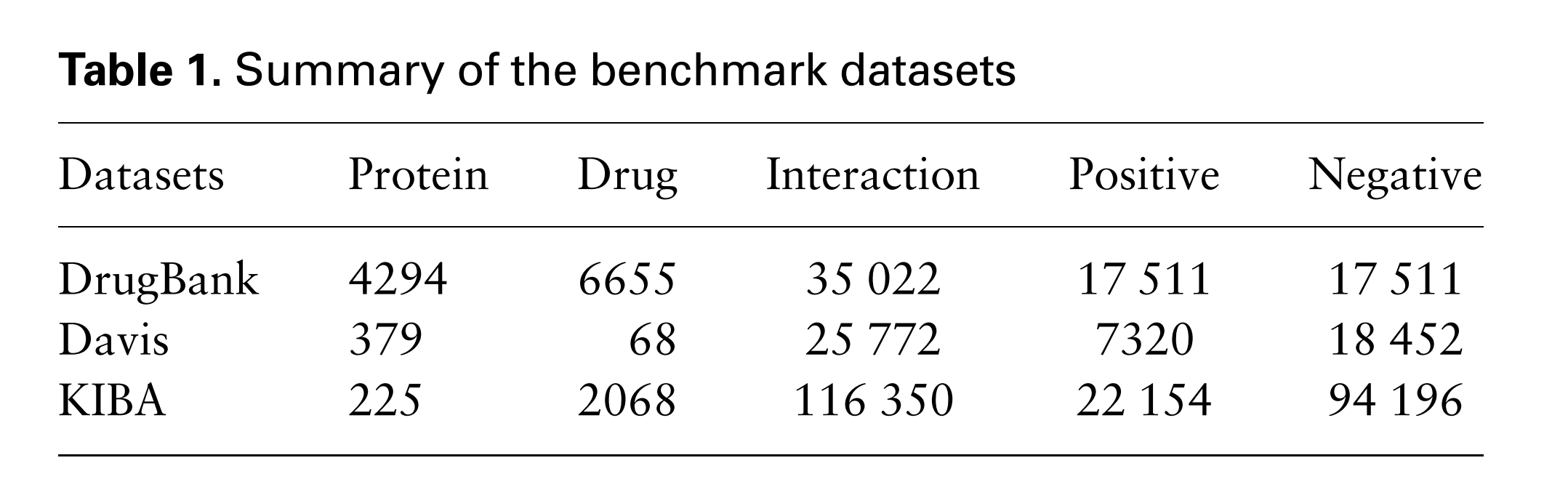
4. BenchMark Datasets : DrugBank, Davis, KIBA
5. Main model architectures :
- Compound : chracterwise Embedding + stacked 1D-CNN
- Protien : chracterwise Embedding + stacked 1D-CNN
- Drug-Protein Cross-Attention
- Global Max Pooling
6. Interaction map idea :
- simple concatenation btw. drug, prot representation vectors + FC Layer
7. Metrics : AUC, PRC, Precision, Accuracy, Recall
8. Task/ Loss function/ optimizer : Classification , BCELoss, AdamW
9. citations (작성일 기준) : 44
간만에 괜찮은 논문을 읽었다. 최근 논문들을 보면 별로 획기적이지 않은 내용을 어렵게만 써서 마치 수준 있는 논문인 것 처럼 둔갑시키려는 얄궂은 짓을 해둔 것들이 많은 것 같다. 그런 논문들에 비하면 이번 논문은 상당히 읽기 쉽게 독자를 배려했으며 비교실험을 직접 돌려보니 실제로 모델의 성능도 좋게 나온다. Sequence Based로 CPI연구를 하는 연구자라면 꼭 비교논문으로 사용해보길 추천한다.
1. Introduction
뻔하지만, Deep Learning을 통해 Drug-TargetProtein Interaction (Compound Protein Interaction)연구가 활발해졌다.
Conv 기반의 DeepConv-DTI, Transformer 기반의 transformerCPI, Moltrans등을 언급, Binding Affinity를 prediction하는 DTA 연구로의 확장에 대한 언급, Graph 기반의 GraphDTA, Attention을 도입한 연구들 등 전반적인 연구 과정 및 모델을 언급
2. Meterials and Methods
- CNN block : protein과 compound의 feature를 learning할 것으로 기대
- Attention block : 얻어진 protein과 compound feature matrices를 input하여 decision vector를 얻는다.
- Output block : Prediction layer
모델 구조 설명, 맨 처음 첨부한 모델 figure 참고
- Drug와 Protein을 위한 각각의 independent CNN block이 존재함
- 각 1D CNN은 protein(P_cnn)과 Drug(D_cnn)의 feature vector를 학습 및 생성
- CNN block의 output인 P_cnn은 M x f, D_cnn은 N x f의 size를 가지는 matrix이 된다.
-
- 여기서 M과 N은 protein/drug max sequence length
- f는 cnn의 last layer output channel 수인데, 그냥 feature vector의 dimension으로 생각하자.
- 따라서 각 시퀀스들(p1, p2, ... ,pM / drug는 d1, d2, ... ,dN)은 f x 1 size를 가진다.
Attention block :

정리하면 : Dcnn과 Pcnn을 가지고 서로에 대해 attention된 attention matrix A를 만들겠다는 것, A의 size는 N x M x f이다.
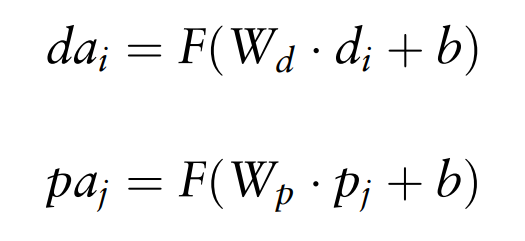
-
F는 activation function
-
Pcnn은 M x f , 각 시퀀스 하나(pj)는 f x 1 의 사이즈를 가진다.
-
Dcnn역시 N x f 로써, 각 시퀀스 하나(di)는 f x 1 의 사이즈를 가진다.
-
여기에 각각 f x f size의 learnable matrix Wp 와 Wd를 곱하면 f x 1 의 벡터를 얻게 된다, (dai, paj)
-
bias 역시 f x 1 의 사이즈를 갖는다.
-
이제 drug의 i번째 element와 protein의 j번째 element의 Attention vector A(i,j)를 얻기 위해서 다음의 식을 계산한다.
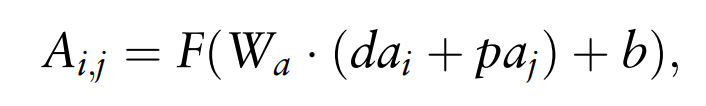
-
dai + paj는 elemental wise adding이므로 f x 1 의 사이즈를 그대로 가진다.
-
bias (f x 1)과 더하는 부분이 있기 때문에 Wa matrix와의 연산 결과 역시 f x 1 이 되어야 한다.
-
따라서 Wa 사이즈는 f x f 이다.
-
결과적으로 drug의 i번째 시퀀스와 protein의 j번째 시퀀스의 attention vector는 f x 1 벡터이다.
-
그러면 i는 Drug의 element이므로 결국 1, 2, 3... , N까지 존재하고 j는 M개 까지 존재한다.
-
모든 i와 j에 대해 위와 같은 연산을 진행하고 나면 자연스럽게 얻어진 Attention Matrix의 size는 M x N x f가 된다.
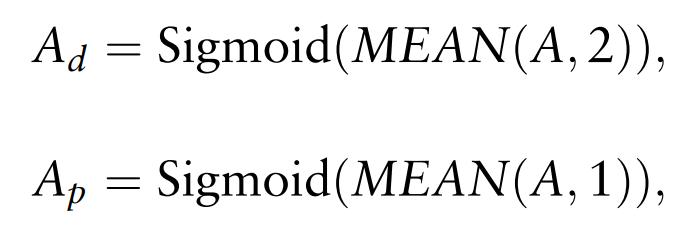
-
다음으로 drug와 protein의 Attentioned matrix를 얻기 위해 위 연산을 진행한다. 그냥 각 방향에 대해 global average를 취해주면 된다.
-
Ad는 N x f Ap는 M x f 의 사이즈를 갖게 된다.

-
마지막으로 Drug와 Protein의 Latent vector를 얻기 위해 다음과 같은 연산을 진행한다. 기호는 elemental wise multiplier
-
당연히 Da와 Pa의 size는 변동 없이 각각 Ad, Ap와 같다. (Da = RN x f, Pa = RM x f)
마지막으로 Da와 Pa에 대해 Global Max Pooling을 통해서 두 벡터의 사이즈를 f x 1 으로 축소한다.
최종적으로 vd, vp가 얻어지고 concatenated해서 FCNN로 연결한다.
Output block : FCNN을 이용해 output shape 2로 연결시키고 prediction, BCE Loss를 이용한다.
데이터의 Active/Inactive ratio에 반비례해서 데이터가 적은 class에 대해서는 Loss에 대한 가중치를 크게 줘서 class imbalance를 회피하는 전략을 사용했다.
3. Experiments and results
실험은 총 4가지 모드로 진행
-
E1 : train/test 데이터의 중복을 따지지않고 random split
-
E2 : train 데이터에 사용된 drug는 test set에는 존재하지 않음
-
E3 : train 데이터에 사용된 protein은 test set에는 존재하지 않음
-
E4 : train 데이터에 사용된 drug와 protein 모두 test set에는 존재하지 않음
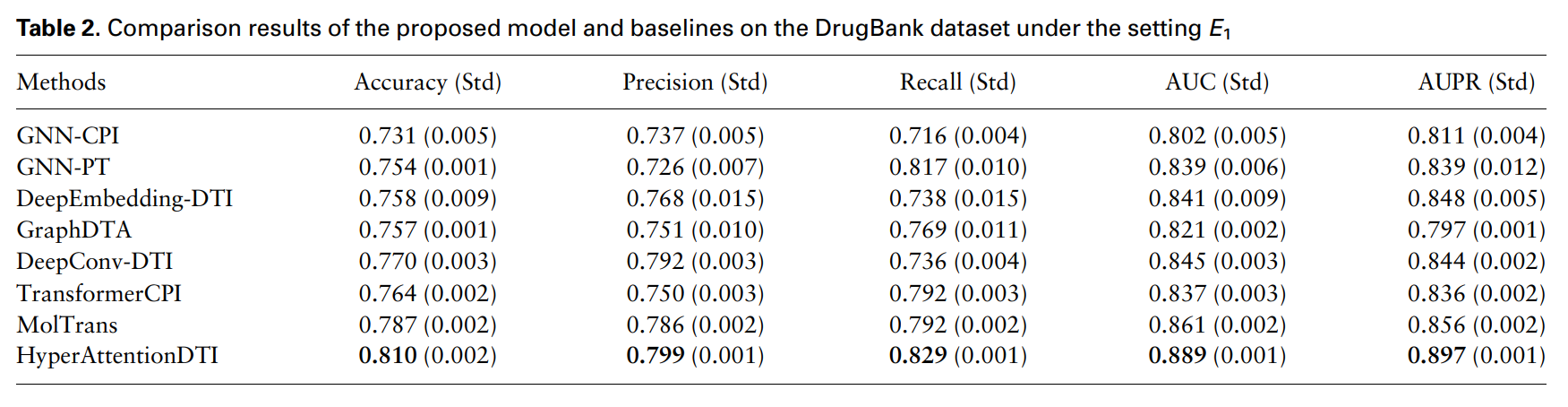
The effectiveness of attention block :
-
No_Attention_DTI
-
Attention_DTI
-
multi-head attention
Case studies (이부분이 주목할 실험부분)
DrugBank데이터로 학습된 모델에 GABAR protein의 16가지 sub proteins에 대해서 drug bank origniated compounds 6708개를 input.
( 6708개의 compound는 DrugBank origin만 같을 뿐 DrugBank데이터를 이용한 train / test에서 사용된 데이터와는 별개의 데이터임)
(다만, train 및 test 데이터셋 자체에도 각 protein과 interaction이 있는 compound들이 섞여있기는 함, 완벽하게 fair한 실험을 하려 했다면 train / test에서 GABAR 16개 sub protein에 대해 interaction이 있는 것으로 알려진 compound는 제거하고 학습한 모델로 실험하는 게 맞다고 봄, 약간의 컨닝 페이퍼가 되지 않나 싶음)
- 6708개의 compound에 대해서 각각의 protein에 대해 interaction probability를 가지고 Ranking을 정함.
- 상위 10개의 compound를 얻고, 그 compound의 DrugBank ID를 통해서 DataBase에 실제로 interaction이 있는 drug로 등록되어있는지를 확인
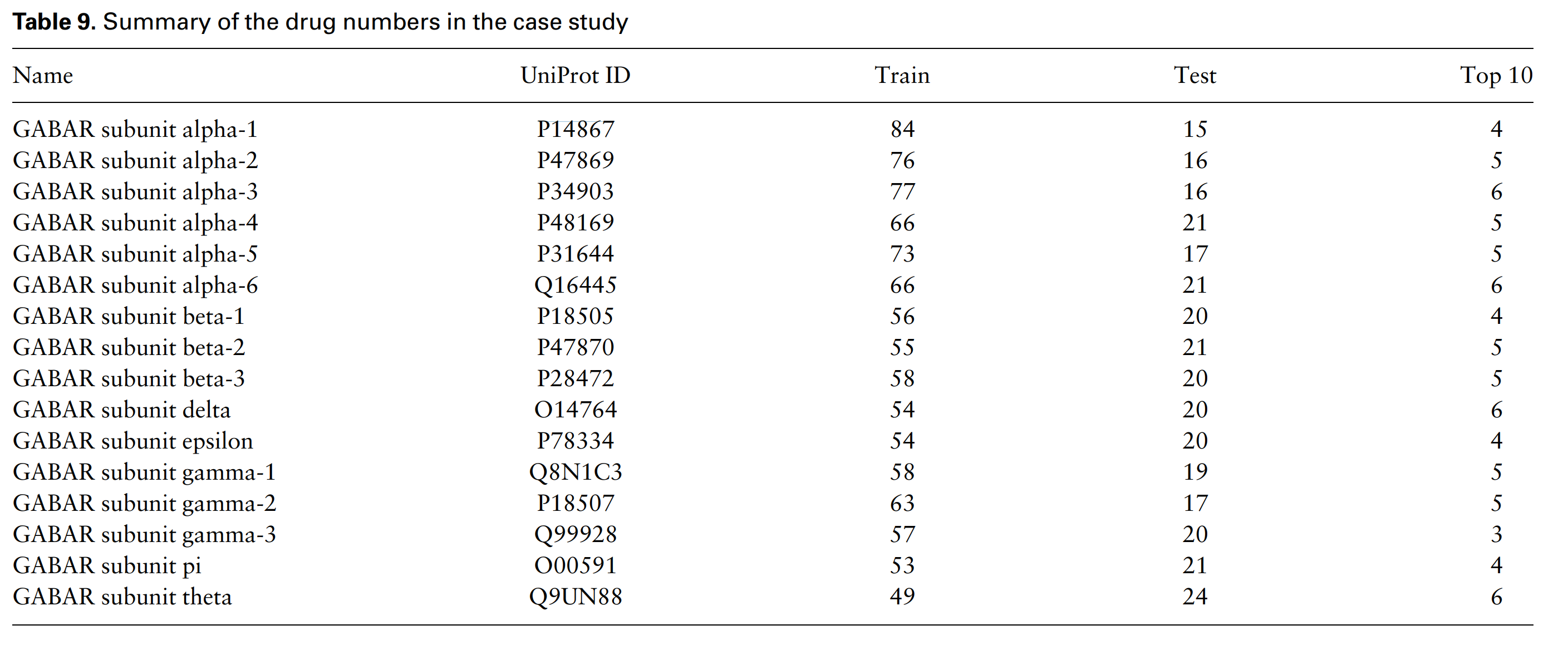
맨 오른쪽 Top10 column은 top10 rank에서 DrugBank에 interaction이 있다고 등록된 Compound의 숫자를 의미한다. 다시말해 모델이 target protein과 interaction할 것으로 예측한 상위 10개의 compound들 중, 실제로 DrugBank에 해당 compound들이 target protein과 interaction이 있는 것으로 등록된 것들의 개수. 나름 반타작 정도는 한다.
- 비슷한 상위 10개의 compound끼리는 structural similarity까지 보여서 visualization까지 해보았다고 함.

- 빨간색이 실제로 GABAR target으로 FDA 승인을 받은 compound의 ECFP를 t-SNE 차원축소한 것,
- 초록색은 모델이 실제로 효과가 있을 것으로 예측한 compound들
-
파란색은 모델이 효과가 없을 것으로 예측한 compound들
다만, 위 실험은 아쉬운점이 Top10 ranked들의 dot만 표시한 figure도 있어야한다고 생각함
Model interpretation
smiles, protein sequence를 이용해 모델을 학습시킨다. 마지막에 protein Attention Matrix Ap (M x f)를 return한다.
Ap를 f방향으로 average pooling 하여 (M x 1) 사이즈의 1차원 score 벡터로 만든다. 해당 score 벡터는 protein sequence의 length(M)과 사이즈가 같기 때문에 각 protein에 대해 attention score를 mapping할 수 있다.
얻어진 벡터의 값을 이용해서 protein Sequence에 score를 mapping한 뒤 3D visualization.
-
Crystal structure of HIV protease D545701 bound with GW0385 (PDB: 2FDD)
-
Crystal structure of type 2 PDF from Streptococcus agalactiae in complex with inhibitor AT018 (PDB: 5JF3).
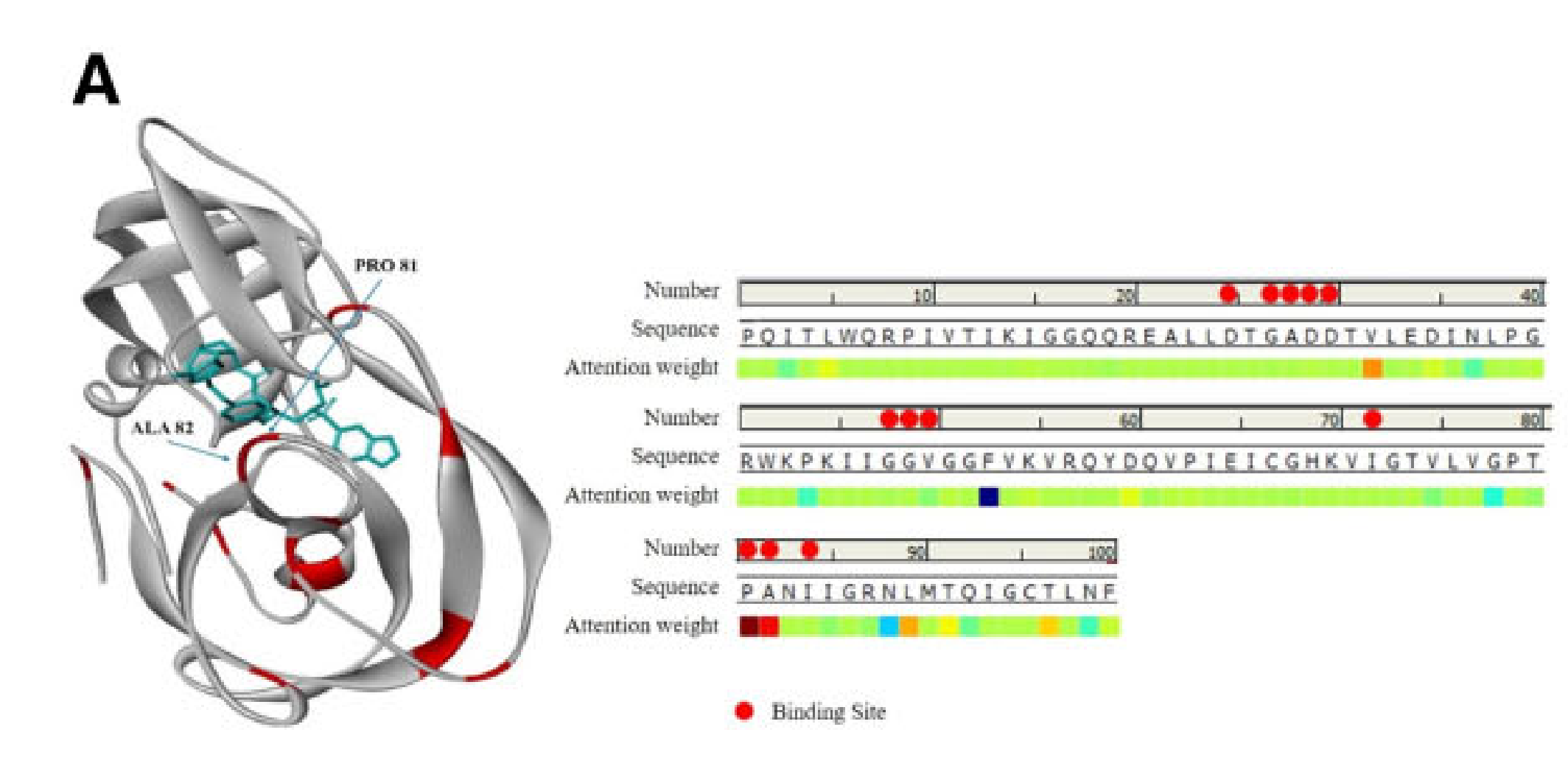
실제로 compound와의 binding site에서 attention score가 높은 경우를 발견,
그러나 binding site가 전혀 아닌데도 높은 attention score를 가지는 것도 많이 있다고 함.
해당 논문에서 얻게된 insight 정리
- 기존에 사용하던 instance based row resolution attention mechanism에서 feature based high resolution attention mechanism을 사용한 점은 상당히 뛰어난 전략같다.
- Case study들이 상당히 현실적이고 매력적이었다.
'Paper > Bioinformatics' 카테고리의 다른 글
| [2022 NIPS] DIffAb 논문리뷰 (1) (0) | 2025.03.27 |
|---|
- Total
- Today
- Yesterday
- manimtutorial
- eigenvector
- manim library
- 기계학습
- nanobody
- kl divergence
- ai신약개발
- 나노바디
- 오일석기계학습
- 3B1B따라잡기
- elementry matrix
- eigenvalue
- 베이즈정리
- 이왜안
- manim
- 인공지능
- kld
- MorganCircularfingerprint
- MLE
- Matrix algebra
- 3b1b
- 백준
- 제한볼츠만머신
- marginal likelihood
- Manimlibrary
- 파이썬
- MatrixAlgebra
- 최대우도추정
- 선형대수
- variational autoencoder
| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |
| 29 | 30 |
