티스토리 뷰
내가 다루는 데이터는 Target Protein - SMILES seq - pKa Value로 구성된 triplet structure의 dataframe이다.
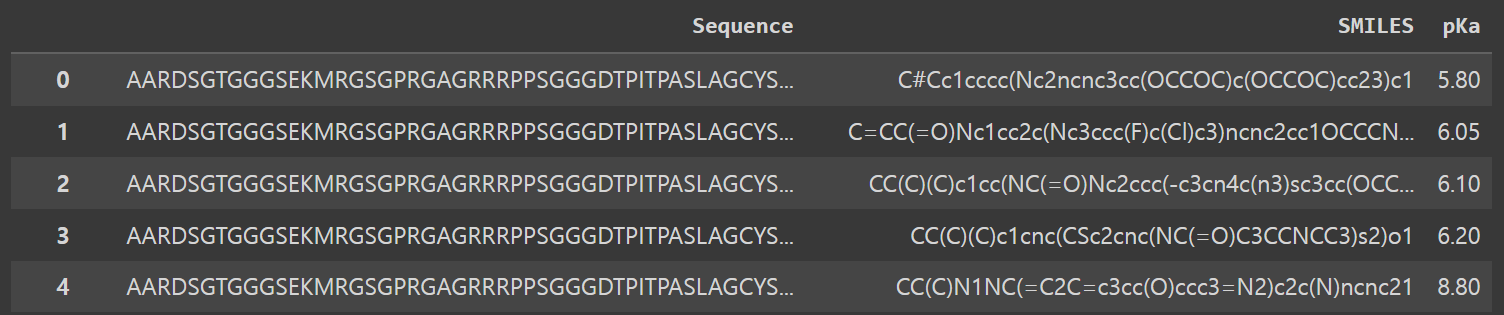
그런데 문득 그런 생각이 들었다. 내가 공부를 하면서 주로 Y 값(여기서는 pKa)으로는 Kd Ki 또는 IC50 EC50 value를 사용하는데 왜 내가 가진 데이터는 pKa값을 사용하는가?
나는 pKa값을 어떤 물질의 산성도를 나타내는 척도? 정도로 사용된다는 애매모호한 값으로만 기억하고 있었다, 부끄러운 일이지만 실제 생물 실험방에서는 pH 미터기를 사용하지 pKa 미터기를 사용하는 일이 없었기 때문에 pH에 대해서는 기억하더라도 pKa를 기억하고 있을 이유가 없었기 때문이다.
그러나 Biological CS분야의 연구에서 이렇게 Drug-Target seq를 이용해 Y값을 regression하거나 classification 하는 경우가 상당히 많은데, 이때 위에서 언급한 것 처럼 pKa, Kd, Ki, IC50, EC50 등의 다양한 값을 사용하기 때문에, 이 값들의 의미를 모르면 어떤 값의 threshold를 설정함에 있어서 상당한 혼란이 있을 것이라 생각해서 간단하게 pKa, Kd, Ki, IC50, EC50 에 대해 개념을 정리해보려 한다.
1. pH (power of Hydrogen)
pH값은 용액속에 자유롭게 떠돌아다니는 H+농도라고 생각하면 된다.
이때 pH value는 -log[H+]로 나타내며 [H+]값은 수소 이온의 농도이다. p의 의미는 -log10로 생각하면 된다.
예를 들어 물에는 1/107의 농도로 H+이온이 존재한다.
따라서 [H+] = 1/107 = 1 x 10-7 이므로 -log[H+] = -log(1 x 10-7) = 7이므로 물의 pH 값은 7이 된다.
기본적으로 pH7값을 중성으로 잡으며, 이보다 작은 물질을 산성, 큰 물질을 염기성이라고 배웠던 것은 기억할 것이다.
아래의 명제들을 차근차근 생각해보자.
a) 물은 중성이며, [H+] 농도는 1/107이고 이때 pH값은 7이다.
b) 물보다 pH값이 작은 물질은 '산성'이며 pH값이 7보다 작아야 하므로 [H+]농도는 1/107보다 높아야한다.
(다시말해, 용액속에 H+의 양이 많다. <---> 용액속에 OH-의 양이 적다. == 산성이다.)
c) 반대로 물보다 pH값이 큰 물질은 '염기성'이며 pH값이 7보다 커야 하므로 [H+]농도가 1/107보다도 낮아야한다. (다시말해, 용액속에 H+의 양이 적다. <---> 용액속에 OH-의 양이 많다. == 염기성이다.)
2. Ka and pKa
Ka 는 두가지 지표로 사용되는 것 같다.
하나는 Acid Dissociation constant이다. 용액에서 산성도를 양적으로 측정하는 척도가 되어준다.
pKa는 pH에서와 마찬가지로 -log10Ka로 생각하면 된다.

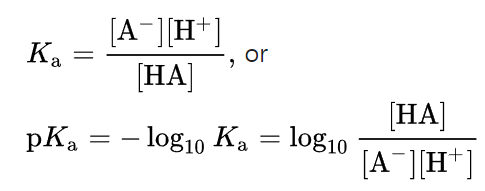
차근히 생각해보자, 오른쪽 식을 살펴보면 Ka값이 높다는 것은 물질속에 HA로 결합된 상태보다 A-와 H+로 해리되어 있는 상태의 물질이 더 많다는 것을 의미한다. 물질속에 H+가 많다는 것은 pH가 매우 낮다는 것을 의미하고, 산성임을 의미한다. 따라서,
a) Ka 값이 클수록 해당 물질은 산성도가 높다. <---> Ka값이 작을수록 해당 물질은 산성도가 낮다.
b) pKa값은 pH와 똑같이 적용된다. 따라서 pKa값은 작을수록 산성도가 높고 클수록 산성도가 낮다.
결국 이때의 pKa는 pH와 똑같이 물질의 산성도를 나타내는 척도라고 이해하고 있으면 된다.
두번째로, 아마 이것이 더 많이 이용되는 경우라고 생각되는데 Association constant를 의미한다.
Association constant는 아래에서 설명할 Kd의 역수이다. 그냥 정말 직관적으로 Kd와 정 반대의 의미를 가지는 역수값이라 생각하면 되고, 의미로는 [PL] / [P][L] 이다. 아래에서 Kd의 개념을 설명하니 먼저 Kd 설명을 읽고 와서 반대로만 생각해주면 된다.
3. Kd vs Ki
일단 Kd Ki value에 대해 설명하기 전에 Ligand라는 용어는 Protein단백질과 결합하는 상대적으로 작은 분자량을 가지는 기질들의 집합을 ligand라고 생각하면 된다.
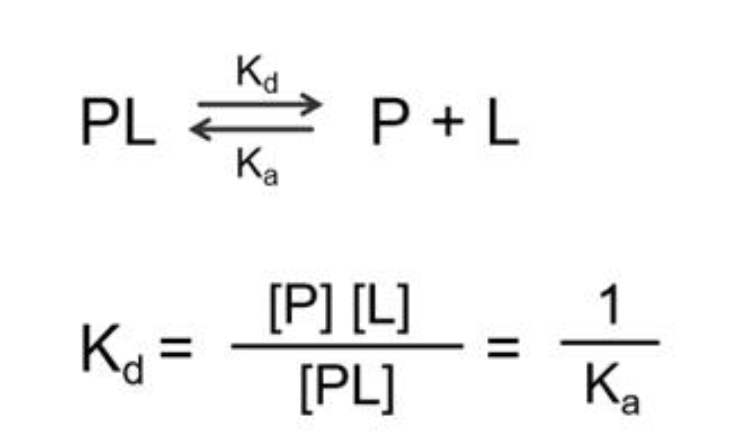
Kd와 Ki는 위에서 언급한 pKa와 pH와는 다르게 Ligand와 Protein또는 Drug, Target등의 Binding Affinity를 나타내는 척도로 사용된다. Kd는 Dissociation constant이며 Ki는 Inhibition constant이다.
여기서 PL = Protein-Ligand , P = Protein , L = Ligand이며 [ ] 표시는 마찬가지로 농도를 나타낸다.
Dissociation된 [P][L] 값이 크고 association된 [PL]값이 작을수록 Kd값이 커질 것이라고 생각할 수 있다.
또한 [P][L](Dissociation rate)의 의미는 Protein과 Ligand가 서로 분리되어 존재하는 농도를 의미한다. [PL](Association rate)의 의미는 Protein과 Ligand가 결합한 채로 존재하는 농도를 의미한다. 상식적으로 생각하면 된다. Protein과 Ligand가 결합력이 강하다면 [PL]형태로 더 많이 존재할 것이고 [P][L]형태로는 적게 존재할 것이다. and vice versa. (뜬금없지만 알아두면 좋은 표현이므로 무슨말인지 모르면 검색해보길 추천)
즉, Kd값이 클수록 [P]와 [L]의 Binding Affinity는 작다, 반대로 Ka값은 클수록 Binding Affinity가 크다는 것을 알 수 있다.
a) Kd값이 크다는 것은 Protein-Ligand사이의 Binding affinity가 낮다라는 것을 의미한다.
b) 반대로 Kd값이 작다는 것은 Binding affinity가 높다라는 것을 의미한다.
Ki값 역시 dissociation constant와 비슷한 값이지만, 특히 Protein Ligand의 관계가 inhibitor로써 작용할 때 사용하는, Kd보다 narrow한 의미의 dissociation constant라고 생각하면 된다. 즉 Ki값 역시 Kd와 똑같이 크면 Binding affinity가 낮고, 더 많은양의 inhibitor가 있어야 inhibition 작용을 나타낼 수 있다고 이해해야 하며, 작으면 Binding affinity가 높고, 적은 양의 inhibitor로도 충분한 inhibition 작용을 나타낼 수 있다고 이해하면 된다.
4. IC50
IC50는 Inhibitory concentration 50%라는 말인데, 어느만큼의 농도를 처리했을 때 inhibition rate가 50%를 달성하느냐를 의미하는 것이다.
a) IC50값이 작을수록 inhibitor의 binding affinity가 높다는 것을 의미한다.
b) IC50값이 클수록 inhibitor의 binding affinity가 낮다는 것을 의미한다.
다만 Ki나 Kd처럼 직접 농도를 가지고 계산하지 않으므로 상대적으로 정확성이 떨어지며 inhibition mechanism과 측정 조건에 따라 상당히 다른 값이 얻어질 수 있어서 주의해야한다. 계산식은 약간 복잡하지만 컴퓨터 분야로 옮겨온 이상 IC50나 EC50의 정확한 계산 방식까지 공부할 필요성은 없다고 생각하므로 여기서는 생략한다.
5. EC50
EC50은 Effective concentration 50%라는 말이고 maximum effect의 50%를 달성하는 농도를 의미한다. 이것도 자연스럽게 낮을수록 더 적은 농도의 물질로도 더 좋은 효과를 나타냄을 의미한다.
a) EC50값이 작을수록 drug의 binding affinity가 높다는 것을 의미한다.
b) EC50값이 클수록 drug의 binding affinity가 낮다는 것을 의미한다.
마찬가지로 IC50처럼 정확하게 계산하는 방식들이 있지만 여기서는 생략한다.
↓ 내용이 혹시나 도움되셨다면 눌러주세요 꾸준한 포스팅에 큰 원동력이 됩니다 🥰
'Background > Biology' 카테고리의 다른 글
| 나노바디 (Nanobody)리뷰 (1) 구조적 특징 (0) | 2025.02.03 |
|---|---|
| Antibody Antigen Generative Modeling (3) Modularity (0) | 2025.01.31 |
| Antibody Antigen Generative Modeling (2) Learnability (0) | 2025.01.28 |
| Antibody Antigen Generative Modeling (1) Introduction (0) | 2025.01.23 |
| Morgan fingerprint, Morgan circular fingerprint? (4) | 2023.04.16 |
- Total
- Today
- Yesterday
- 나노바디
- 3B1B따라잡기
- ai신약개발
- marginal likelihood
- 3b1b
- 최대우도추정
- 인공지능
- eigenvalue
- MLE
- 논문리뷰
- Matrix algebra
- 백준
- nanobody
- elementry matrix
- 파이썬
- MatrixAlgebra
- eigenvector
- 선형대수
- Manimlibrary
- manim
- antigen antibody interaction prediction
- manimtutorial
- 항원항체결합예측모델
- dataloader
- MorganCircularfingerprint
- manim library
- 오일석기계학습
- 베이즈정리
- 이왜안
- 기계학습
| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | ||||
| 4 | 5 | 6 | 7 | 8 | 9 | 10 |
| 11 | 12 | 13 | 14 | 15 | 16 | 17 |
| 18 | 19 | 20 | 21 | 22 | 23 | 24 |
| 25 | 26 | 27 | 28 | 29 | 30 | 31 |
