티스토리 뷰
https://biomadscientist.tistory.com/177
HuDiff/HuDiff-Nb 논문 리뷰 (1) Introduction
https://www.biorxiv.org/content/10.1101/2024.10.22.619416v1.fullhttps://github.com/TencentAI4S/HuDiffHuDiff는 기존 CDR 서열 생성에 치중하고 있는 모델들과 달리 오히려 Framework Regions(FRs)의 서열 생성을 통한 인간화 (Humani
biomadscientist.tistory.com
Architectures of HuDiff
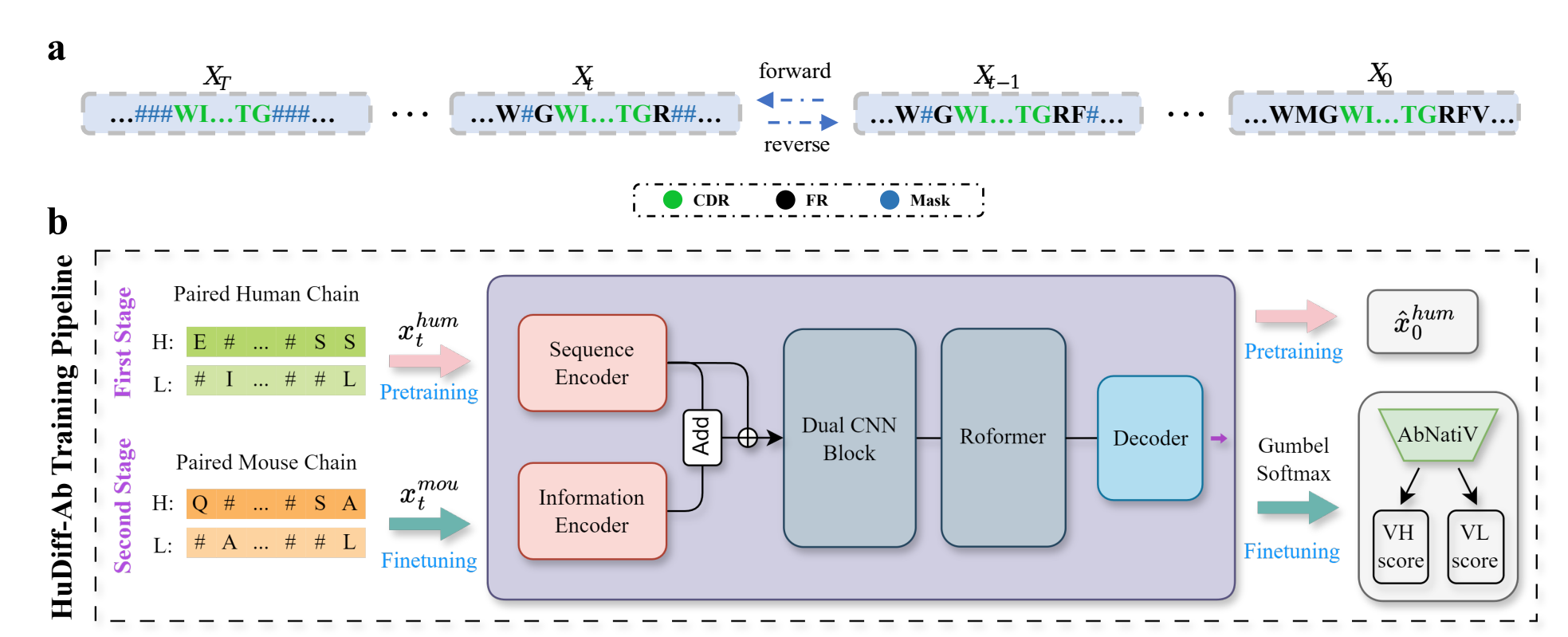
HuDiff-Ab
Input :
[Pre-training] Human paired, aligned HeavyChain(HC), LightChain(LC) 서열
[Fine-tuning] Mouse paired, aligned HeavyChain(HC), LightChain(LC) 서열
Align : IMGT numbering Method를 사용했고 HC은 152, LC는 139로 Length를 맞춰서 Alignment진행
Token : '-' Padding token, '#' as Diffused state token(완전히 Noising된 상태를 말하는 듯), 'X' as Unknown residue type 따라서 토큰의 갯수는 23개로 고정함 (일반적인 아미노산 서열 20 + 3)
[WorkFlow : Pretraining]
1. Input된 Aligned Sequences는 Diffusion step 0~T를 따라서 RF 서열들이 #로 various하게 Masking됨
➡️ 2. Tokenizer에 의해 Numerical vector로 변환됨 (Sequence Encoder)
➡️ 3. Extra Information Encoder를 이용해서 Dual CNN Block과 동일한 shape를 가지는 학습된 representation vector 반환
➡️ 4. Add + Concat한 representation vector를 HC과 LC따로 나눠서 학습하는 1D CNN Block에 의해 학습된 representation vector 반환 (Dual CNN Block)
➡️ 5. Sequence dependency를 학습하는 Rofomer (self-attention block)
➡️ 6. MLP Decoder를 이용해서 원본서열을 return
➡️ 7. Output 원본서열은 Gumbel-softmax를 이용해서 one-hot vector로 encoding
➡️ 8. AbNativ를 이용해서 VH와 VL의 socre를 계산하여 모델을 평가하고 학습
[WorkFlow : Finetuning]
* Input만 Mouse의 것으로 사용하고 그 외는 동일
[Objective Function]
Autoregressive diffusion method (DDDPM 참고)
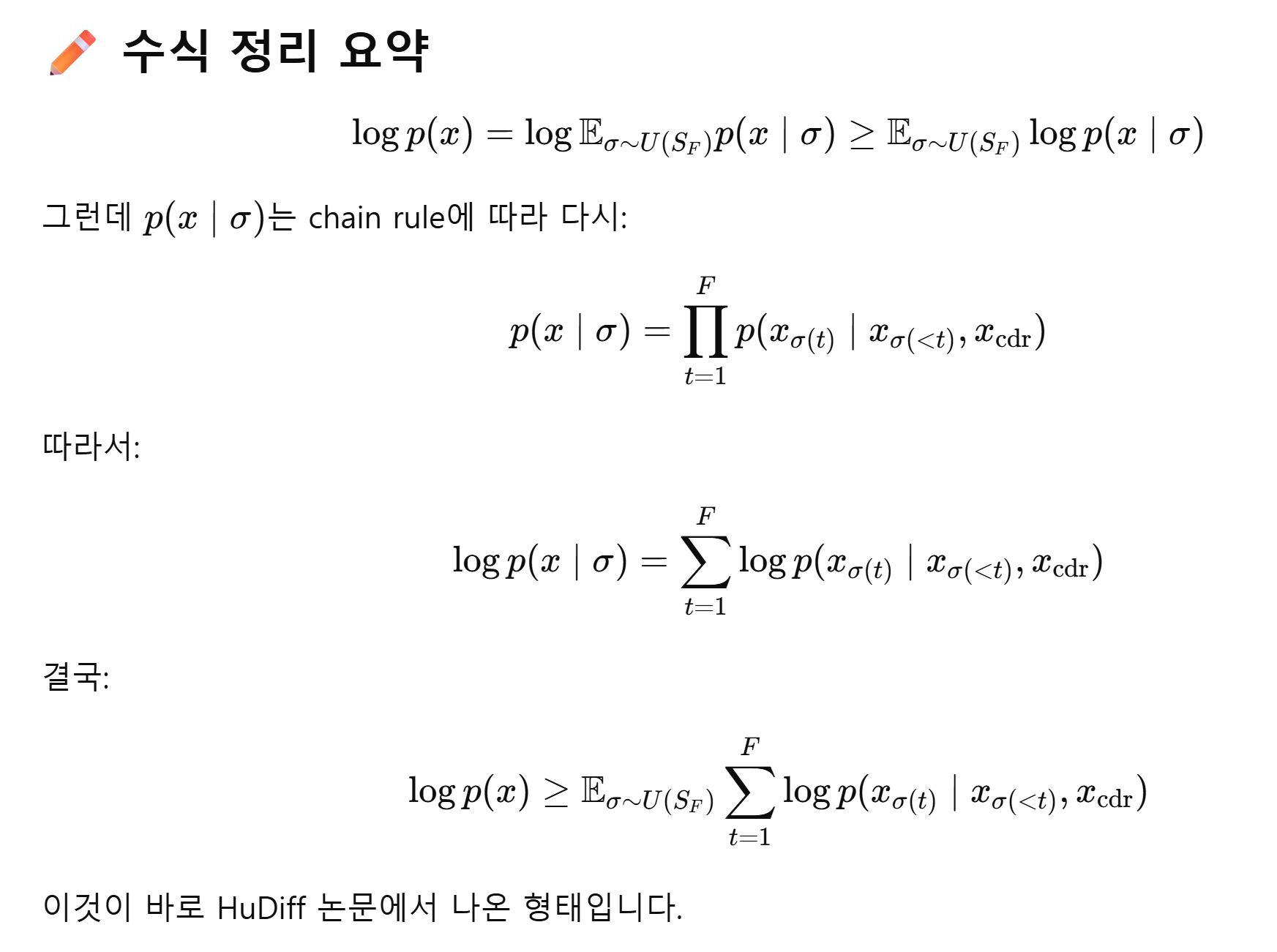
D3PM은 transition matrix를 이용해서 원본서열을 복원하는 학습방법으로 기존 autoregressive model의 경우 chain rule에 의해서 데이터 x의 발생할 log-likelihood를 다음과 같이 정의함

그러나 HuDiff는 MLM을 이용해서 서열을 직접 복원한다고 함. 그리고 이 논문에서는 중요하게 보는 포인트를 완성된 문자열 p(x)의 log-likelihood를 맨 처음부터 순차적으로 p(x1|x0), p(x2|x0,x1), ... ,p(xT|x0,x1,...xT-1) 형태로 구하지 않고 order-agnostic autoregressive model(이하 OAAM)로 구현하는 것을 생각한 것 같음. 이것을 위해 FR의 서열들을 무작위로 선별하기 위한 순열세트를 만들었음
- pre-training 동안 랜덤서열 σ가 SF 집합으로부터 uniformly drawn됨
- SF는 1~F 까지의 가능한 모든 순열의 조합들을 원소로 가지는 집합.
- ex) {{1, 3, F, 32, ...}, {3, 18, 21, ...}, ... ,{모든 가능한 1~F를 이용한 순열들}},
- F는 FR의 길이
Equation 1.을 본 모델의 OAAM log-likelihood로 적용하면 Jensen 부등식에 의해서 다음과 같은 식을 얻을 수 있음.

log함수는 concave함수이므로 젠슨(또는 얀센) 부등식을 적용하면 다음식을 항상 만족한다.
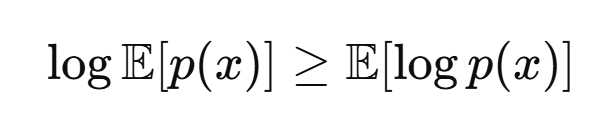
그리고 σ에 대한 Expectation 식은 너무 어렵게 생각하지 말고 직관적으로만 이해하면 된다. 문자열을 맨 처음부터 순서대로 학습해서 최종 문자열의 log likelihood를 학습하는 것 대신 예측 순서를 랜덤하게 한번 뒤섞은다음 최종 문자열을 예측하도록 하는 것이다.
예를들어 KMSLWQL 이라는 단백질 서열이 있다고 하자, 기존의 Autoregressive Model의 학습 방식은 K_______ 정보를 준 뒤 K'M'______을 예측하도록 하고, 다음으로 KM'S'_____를, 이렇게 순차적으로 예측하여 마지막으로 KMSLWQ'L'을 예측하는 방식이라면 OAAM은 직관적으로 이러한 방식이다. ___'L'___ -> _'M'_L___ -> _M_L'W'__ -> _M_LW'Q'_ -> ... -> KMSLWQL
이것을 수식적으로 풀어둔 것이 Equation 2.이다.
Summation기호는 t=1~F까지 순서대로 해당 token의 log likelihood를 말그대로 문자열의 길이 F개까지 summation하는 것이고, 조건부로 붙어있는 xσ(<t)는 예시에서 설명 한 것 처럼 t이전까지만의 서열의 정보를 입력으로 사용한다는 것, xcdr은 모델이 뱉어내는 log likelihood값을 얻기 위해 모델에 cdr의 정보를 함께 전달한다는 의미이다.
그렇다면 왜 이런 OAAM Objective Function을 제안했을까?
- 일반적인 autoregressive 모델은 고정된 순서 (예: left-to-right) 로 학습함
- 하지만 diffusion 모델 (특히 HuDiff와 같은 D3PM 스타일)은 masking 순서를 매 step 랜덤하게 뽑음.
- 따라서 모델이 모든 가능한 순서에 대해 robust하게 학습되도록 하려면, 여러 순서를 평균한 objective가 필요함
→ 이 평균된 loss를 최적화하면 전체 log likelihood에 대한 lower bound를 최대화하는 셈
ChatGPT를 이용해서 잘 정리된 수식이 있으니 참고
어쨋든 Equation 2.는 다시 아래와 같이 re-weighted trick을 사용해서 변형해준다.
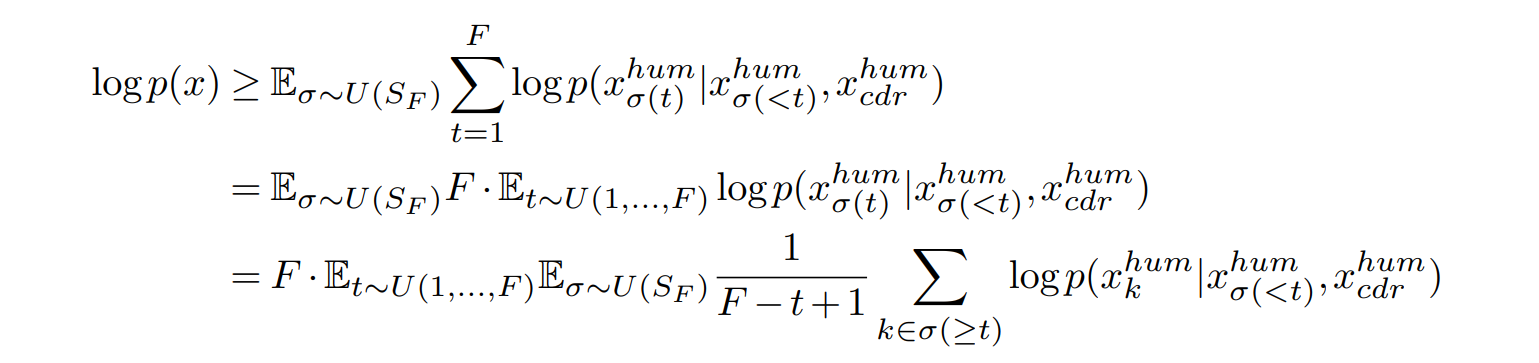
근데 이 re-weight trick이 이해하기가 조금 난해할 수 있다. 너무 수학적으로 까다롭게 증명하려 하지 말고 직관적으로만 이해하자.
σ와 t를 하나씩 뽑아서 log likelihood를 계산하는 대신 학습 안정성을 높이기 위해 t시점 이후의 모든 가능한 position k∈σ(≥t)에 대해 log-probability를 평균 내는 방식으로 학습을 구성함.
이런 의미를 수식으로 표현한 것인데, 쉽게생각하면 우리가 딥러닝 모델을 학습시킬 때 보통 하나의 데이터를 넣어서 online-learning을 시키지 않고 여러개의 데이터를 batch size 단위로 넣어서 그 데이터들로부터 얻어진 Loss값을 평균으로 model을 backpropagation하는 batch learning을 하는 경우가 정석인 것은 이제 너무 자명한데, 그걸 log-likelihood 관점으로 적용한 것으로 생각하면 된다.
Equation 2.처럼 서열 하나마다 계산되는 log-likelihood를 계산하고 Loss로 주는 것은 Variance가 커서 학습 안정성과 관련해서 별로 좋지 못한 방법이라는 판단 하에 저자들은 '어차피 σ와 t가 Uniform distribution이니까 임의의 position k이 선택될 확률은 1/F로 동일하므로, 선택된 임의의 position k보다 후방에 위치한 예측해야되는 모든 후보 서열 σ(≥t) 들의 log-likelihood의 평균 값을 Objective Function으로 구성하자'고 제안하는 것이다.
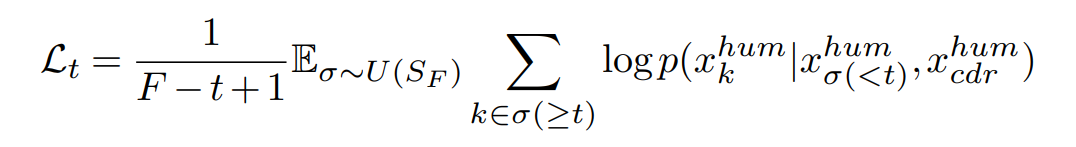

이제 Equation 4.에서 적절한 하나의 term을 Lt로 치환해주면 다음과 같은 완성된 ELBO를 얻을 수 있다. 반복적으로 t를 Uniform Distribution에서 샘플링하고 F*Lt를 계산하여 Loss로 사용하면 ELBO term을 Maximization 할 수 있다.
[Pretraining Loss Term]

Pre-training 단계의 최종 Loss Term은 다음과 같고 Lassistant는 input된 CDR의 서열과 output CDR 서열이 동일한지를 기반으로 설정되는 Assistant 형질의 loss라고 한다. (? 모델 구조상 CDR은 변하지 않는 것 아닌가? 갑자기 왜 CDR 서열이 동일한지를 Loss로 주는지는 이해가 안가지만.. 대충넘어가자)
Lt는 쉽게말해 원본 FR 서열을 복원하였는지를 확인하는 Reconstruction Loss 라고 생각하면 된다.
[Fine tuning Loss Term]
Fine tuning단계의 Loss는 약간 다르지만 크게 어려울 것 없이 직관적이다.
우선 input되는 서열이 human의 것이 아니라 mouse의 것으로 대체된다. 생성된 서열을 gumbel-softmax를 통해서 one-hot encoding하여 얻어진 xonehot은 RLength of H + L (291)*Token_size(23) shape를 가지는 텐서일 가능성이 높다.
잘은 모르지만 AbNativ는 이런 one-hot 형태의 Residue를 입력받을 수 있을 것으로 생각한다. (AbNativ와 token dictionary의 갯수가 맞을 것 같지는 않은데.. 어떻게 입력되는지는 조금 알아 볼 필요가 있겠다.)
AbNativ는 VHscore와 VLscore를 계산해주고, Lscore에 이 둘 중에서 더 큰 값(max)을 취해서 저장한다.
Pretraining과 마찬가지로 CDR 서열은 원본과 동일하도록 강제하는 Lassistant term을 추가하여 Fine tuning을 진행한다.
HuDiff-Nb는 그냥 HuDiff-Ab를 Nanobody 구조에 맞게 변형만 한 것으로 크게 다르지 않아 간단히 ChatGPT로 요약한 내용으로 남겨놓는다.
🧠 HuDiff-Nb: 나노바디 Humanization을 위한 Adaptive Diffusion 모델 구조 정리
단일 체인으로 구성된 camelid nanobody(VHH)는 작고 안정적인 구조 덕분에 치료제 개발에서 각광받고 있습니다. 하지만 임상 적용을 위해서는 humanization, 즉 인간 친화적인 서열로 변환하는 과정이 필수입니다.
Tencent AI Lab이 제안한 HuDiff-Nb는 diffusion 기반의 autoregressive sequence 생성 방식을 활용하여, template 없이 nanobody를 humanize하는 강력한 프레임워크입니다.
📌 1. HuDiff-Nb의 전체 구조
HuDiff-Nb는 크게 다음과 같은 세 블록으로 구성됩니다:
- ① Sequence Encoder
- Residue token(23종)을 임베딩하여 feature로 변환
- IMGT 기준으로 길이 152까지 padding 처리 - ② Information Encoder
- 포지션 정보, 영역(CDR/FR) 정보 포함
- HuDiff-Ab에는 있었던 체인타입(chain type) 인코더는 단일체인인 VHH에선 생략 - ③ Hidden Block
- CNN Block: 지역적 패턴 학습
- Self-Attention Block: 전체 서열 간 관계 학습 - ④ Decoder (MLP)
- 각 residue에 대해 23차원 logits 예측
- 이후 Gumbel-Softmax로 differentiable one-hot 벡터로 변환
🚀 2. 학습 파이프라인
HuDiff-Nb는 다음 두 단계로 학습됩니다:
✅ (1) Pre-training 단계
- 데이터: human antibody heavy chain (VH)
- 목표: masked FR 영역 복원 → human-like FR 학습
- 학습 방식: autoregressive diffusion 방식으로 timestep에 따라 마스킹된 서열 복원
✅ (2) Fine-tuning 단계
- 데이터: camelid nanobody(VHH)
- 목표: human-like 변환 + camelid 특성(nativeness) 유지
- 출력: Gumbel-Softmax → one-hot 서열
- 평가:
- VH score: human-likeness 평가 (높을수록 인간 항체에 가까움)
- VHH nativeness score: camelid 특성 유지 정도
최종 loss 구성:
Lfinetune = max(VH score) + min(Δ VHH score) + Assistant loss
🧬 Sampling 방식: de vs inp
HuDiff-Nb는 inference 시 다음 두 가지 샘플링 방식을 제공합니다:
| Sampling 방식 | 설명 |
|---|---|
| de (from scratch) | CDR만 입력하고 FR 전체를 샘플링 |
| inp (inpainting) | FR2 영역의 중요 residue는 고정하고 나머지만 샘플링 |
특히 FR2 영역은 발현과 안정성에 영향을 주는 중요한 부분으로, inp 방식은 구조 유지 측면에서 효과적입니다.
🎓 마무리 요약
- Input: VHH sequence (CDR 고정)
- Model: CNN + Self-Attention + MLP decoder
- Output: (152, 23) one-hot sequence
- Loss: VH 점수 최대화, VHH 변화 최소화, CDR 보존 강제
- 특징: 구조 예측 없이 sequence 기반 humanization 가능
📁 관련 리소스
'Paper > CS' 카테고리의 다른 글
| [CVPR 2024] UniBind 리뷰 (0) | 2026.02.13 |
|---|---|
| HuDiff/HuDiff-Nb 논문 리뷰 (1) Introduction (5) | 2025.08.01 |
| Sequence-based prediction of protein binding regions and drug–target interactions(HoTS) 논문 리뷰 (0) | 2023.05.04 |
| DeepConv-DTI 논문 리뷰 (0) | 2023.05.04 |
- Total
- Today
- Yesterday
- MatrixAlgebra
- 인공지능
- Manimlibrary
- elementry matrix
- 백준
- 기계학습
- 오일석기계학습
- 이왜안
- MorganCircularfingerprint
- marginal likelihood
- 선형대수
- 최대우도추정
- dataloader
- 항원항체결합예측모델
- 논문리뷰
- 나노바디
- manim library
- nanobody
- 베이즈정리
- antigen antibody interaction prediction
- eigenvector
- ai신약개발
- 파이썬
- eigenvalue
- manim
- manimtutorial
- MLE
- 3b1b
- 3B1B따라잡기
- Matrix algebra
| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |